 1.- Duplicación del ADN
1.- Duplicación del ADN
La vida de los seres vivos es muy variable , por tanto
para que esta no se extinga ha de haber un momento en se reproduzcan, lo cual lleva
implicito la formación de copias del ADN del progenitor o progenitores .
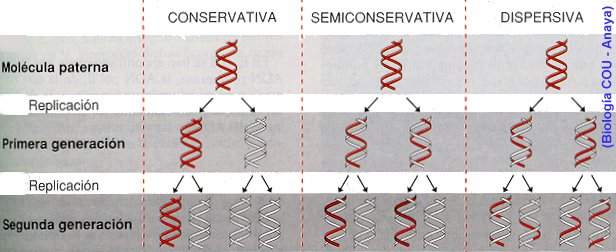
Se dieron
muchas hipótesis sobre como se dupllicaba el ADN hasta que Watson y Crick propusieron la
hipótesis semiconservativa (posteriormente demostrada por Meselson Y Stahl en 1957),
según la cual, las nuevas moléculas de ADN formadas a partir de otra antigua, tienen una
hebra antigua y otra nueva.
MECANISMO DE DUPLICACIÓN DEL ADN EN
PROCARIONTES

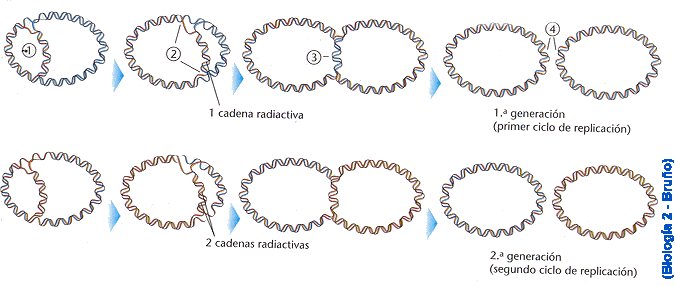
Hay que recordar que es circular y ocurre en tres
etapas:
1ª etapa: desenrrollamiento y apertura de la doble hélice.en el punto ori-c.
Intervienen un grupo de enzimas y proteinas, a cuyo conjunto se denomina replisoma
* Primero: intervienen las helicasas que facilitan en desenrrollamiento
* Segundo: actuan las girasas y topoisomerasas que eliminan la tensión generada por la
torsión en el desenrrollamiento.
* Tercero: Actuan las proteinas SSBP que se unen a las hebras molde para que no vuelva
a enrollarse.
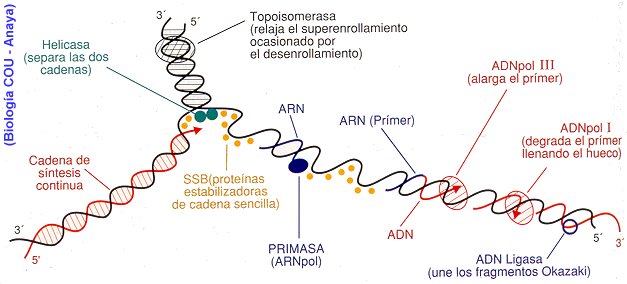
2ª etapa. síntesis de dos nuevas hebras de ADN.
* Actuan las ADN polimerasas para sintetizar las nuevas hebras en sentido 5´-3´, ya que la lectura se hace en el sentido 3´-5´.
* Intervienen las ADN polimerass I y III, que se encargan de la replicación y corrección de errores. La que lleva la mayor parte del trabajo es la ADN polimerasa III
* Actua la ADN polimerasa II, corrigiendo daños causados por agentes físicos.
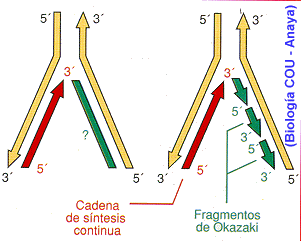
La cadena 3´-5´es leida por la ADN polimerasa III sin ningún tipo de problemas ( cadena conductora). En la cadena 5´-3´ no puede ser leida directamente, esto se soluciona leyendo pequeños fragmentos ( fragmentos de Okazaki ) que crecen en el sentido 5´-3´y que más tarde se unen . Esta es la hebra retardada,llamada de esta forma porque su síntesis es más lenta.
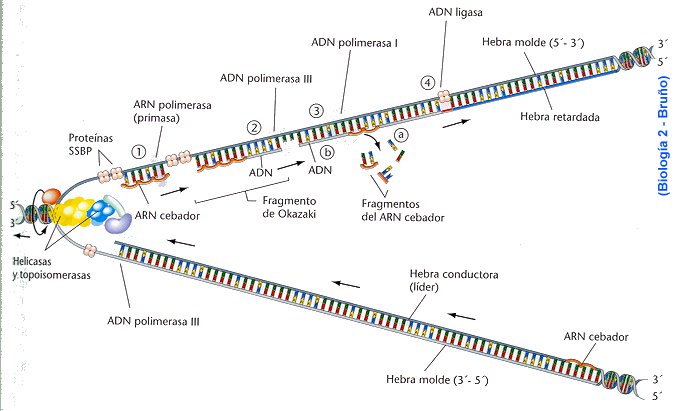
La ADN polimerasa III es incapaz de iniciar la síntesis por sí sola, para esto necesita un cebador (ARN) que es sintetizado por una ARN polimerasa (=primasa). Este cebador es eliminado posteriormente.
3ª etapa: corrección de errrores.
El enzima principal que actua como comadrona (R. Shapiro) es la ADN polimerasa III, que corrige todos los errores cometidos en la replicación o duplicación. Intervienen otros enzimas como:
* Endonucleasas que cortan el segmento erroneo.
* ADN polimerasas I que rellenan correctamente el hueco.
* ADN ligasas que unen los extremos corregidos
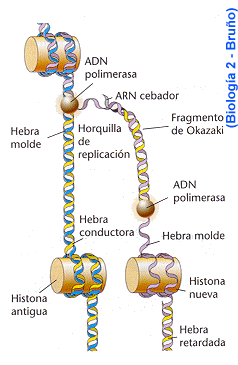
Intervienen enzimas similares a los que actuan en las células procariontes y otros enzimas que han de duplicar las histonas que forman parte de los nucleosomas. Los nucleosomas viejos permanecen en la hebra conductora.

ARTICULO.
El inicio de la replicación del ADN
© Arturo Calzada, Avelino Bueno y Mar Sánchez 2000 abn@gugu.usal.es
RESUMEN
La transmisión fiel
de la información genética de padres a hijos constituye uno de los procesos
celulares más estrechamente controlados y de mayor relevancia para la
reproducción celular. El ácido desoxirribonucléico (ADN) es la molécula
portadora de la información genética de todos los seres vivos y su transmisión
de una célula madre a las células hijas exige el duplicado de esta molécula
¿Cómo dirige una célula la copia de su ADN? Los últimos cinco años de estudios
han revelado que la misma actividad quinasa que controla la progresión por el
ciclo celular activa e inhibe el inicio de la replicación del ADN en una paradoja
tan solo aparente, asegurando de este modo que el material genético sólo se
replica una vez por ciclo celular. Entender como las células regulan la replicación
del ADN, tiene una gran trascendencia no sólo desde el punto de vista del
conocimiento básico, sino también en el diseño de estrategias efectivas para
luchar contra aquellos problemas médicos causados por alteraciones en la
división celular, como el cáncer.
ABSTRACT
The faithful transfer of genome from parents
to progeny is one of the most accurately controlled processes in cell division.
DNA is the molecule bearing genetic information in all living organisms. DNA
has to be duplicated to be transmitted from mother to daughter cells. In
studying problems related to cell division and cancer, it is top priority to
know the processes by which cells precisely regulate initiation of DNA
replication. How do cells drive replication of their DNA?. Studies carried out
during the last 5 years have pointed out
Ciencia al Día © Febrero 2000, Vol. 3, No. 1. ISSN 0717-3849 that the
same kinase activity which controls cell cycle progression, activates and
inhibits the initiation of DNA replication. This apparent paradox ensures that
genome replication precisely takes place only once each cell cycle. In studying
problems related to cell division and cancer, it is crucial to understand how
cells regulate initiation of DNA replication.
El ciclo celular
En 1839, Schleiden y Schwann propusieron la Teoría Celular
estableciendo que cada organismo se componía de una o más células y que cada
nueva célula sólo podía provenir de la división de alguna célula preexistente.
Desde el comienzo de este siglo se sabe que en cada célula, los cromosomas
[Nota 1] portan la información genética. El conocimiento desde los años 50 de
que el material genético es el ADN [Nota 2] y la elucidación de su estructura,
llevó a preguntarse acerca de su replicación. Hacia 1970 se conocía la
composición básica de las células y entoncescomenzaron a abordarse
interrogantes sobre los detalles moleculares de los procesos individuales de la
vida celular.
Las células se reproducen duplicando y repartiendo sus
componentes para constituir dos células hijas. El ciclo celular es el conjunto
de procesos que van desde el momento en que la célula toma la decisión de
dividirse hasta que origina dos células nuevas. Es por tanto la base de la
proliferación celular. Tomando como modelo la levadura Saccharomyces cerevisiae
(S. cerevisiae) revisaremos los conocimientos que se tienen en cuanto a la
regulación de la replicación en las células eucariotas [Nota 3]. Aunque los
detalles de cada ciclo celular eucariota varían entre los organismos, los
puntos esenciales son comunes. En un ciclo celular general (figura 1), para
producir dos células hijas idénticas, el ADN se duplica, se segrega y
finalmente la célula original se divide para dar lugar a dos células nuevas independientes,
cada una con una dotación genética idéntica a la de su hermana.
Los ciclos celulares de la mayoría de las células también
comprenden procesos que conducen a duplicar y dividir su masa y sus orgánulos
citoplásmicos. Por lo tanto el ciclo celular es una compleja combinación de
procesos citoplásmicos y nucleares muy bien coordinados entre sí.
¿Cómo
se organiza el ciclo celular?
El alcance de nuestro
conocimiento del ciclo celular ha sufrido una revolución en los últimos años.
En el pasado, utilizando únicamente el microscopio óptico, los científicos
dividieron el ciclo celular en dos periodos. Uno, de elevada actividad de reorganización
y movimiento de los componentes subcelulares, denominado mitosis, en el que los
cromosomas se condensan haciéndose visibles al microscopio óptico y que
finaliza con los procesos de segregación y división celular. Y un segundo que
comprende la mayor parte del ciclo celular, denominado interfase por transcurrir
entre dos mitosis y en el que a microscopio óptico no se observa proceso activo
alguno salvo el incremento en el tamaño celular. Hoy sabemos que la interfase
no es un periodo de reposo sino que por el contrario es un periodo de gran actividad,
durante el que se llevan a cabo en una secuencia ordenada complicados y elaborados
preparativos para la división celular.
Efectivamente,
durante la interfase y de forma continua tienen lugar acontecimientos conocidos
en su globalidad como crecimiento y que incluyen la síntesis de nuevos
orgánulos celulares y la mayor parte de las proteínas celulares. Además,
comprende procesos (figura 2) que ocurren de forma discontinua, como la replicación
del ADN durante el periodo conocido como fase S -de Síntesis-. El intervalo que
transcurre entre el final de la mitosis del ciclo anterior y el comienzo de la
síntesis de ADN se conoce como fase G1 –del inglés Gap- y el intervalo entre el
fin de la fase S y la mitosis, fase G2.
Las fases G1 y G2
proporcionan tiempo adicional para el crecimiento. Durante G1 la célula analiza
si las condiciones ambientales son adecuadas para comenzar el proceso
irreversible de la división celular, siempre que cuente con el tamaño mínimo
para ello. En el caso de que la situación sea favorable, toma la decisión de atravesar
el punto de Inicio (Start) que compromete irreversiblemente el comienzo del
ciclo celular. Si las condiciones ambientales no son adecuadas para la entrada en
el ciclo, la célula entra en un periodo de quiescencia, denominado G0, en el
que puede permanecer largo tiempo. Además, desde este estadío de G0 la célula
es sensible a señales de diferenciación, en el caso de organismos
pluricelulares, o de inicio de procesos sexuales, en el caso de organismos
unicelulares. Si las condiciones ambientales vuelven a ser favorables, la célula
puede volver a iniciar ciclos de división. En la fase G2 la célula analiza si
ha completadocorrectamente la fase S y decide entre permitir el paso a mitosis,
o en su caso, esperar para que se realicen las reparaciones necesarias.
Control del ciclo
celular
La mayor parte de los
estudios y los primeros avances sobre los mecanismos de control del ciclo
celular en eucariotas se han realizado empleando como modelo las levaduras. Las
levaduras son hongos unicelulares que presentan grandes ventajas como modelo de
estudio del ciclo celular eucariota. Son organismos muy adecuados porque tienen
tiempos de generación cortos, (90 minutos frente a las 24 horas que dura el
ciclo de las células animales) y su genoma es alrededor de 100 veces menos
complejo que el de una célula de mamífero, pero mantienen el mismo tipo de
organización del ciclo celular que los eucariotas multicelulares. Además, es fácil
de manipular genéticamente, siendo posible aislar mutaciones en genes que controlan
procesos celulares básicos, clonar los genes identificados por esasmutaciones y
realizar deleciones [Nota 4] de genes específicos, al ser las levaduras capaces
de vivir como organismos haploides
La mayor parte de los estudios de ciclo celular se han
llevado a cabo empleando S. cerevisiae (Figura 3), la levadura del pan y la cerveza
también conocida como la levadura de gemación por su característico estilo de
división (Figura 4). En el caso de este organismo hay que añadir la ventaja de
que actualmente se conocen los aproximadamente 16 millones de pares de
nucleótidos que constituyen la secuencia completa de su genoma.
Los fundamentos de nuestro conocimiento actual del ciclo
celular en levaduras vienen de la búsqueda sistemática de mutaciones en genes
que codifican para componentes de la maquinaria del ciclo celular realizada por
Hartwell, Culotti y Reid en 1970 en Saccharomyces cerevisiae y Nurse, Thuriaux
y Nasmyth en Schizosaccharomyces pombe (S. pombe) conocida también como
levadura de fisión- en 1976. Basándose en que la división celular es esencial
para la proliferación de estos organismos, aislaron mutaciones que afectan al
ciclo celular como mutaciones condicionales: inactivan un producto génico en
unas condiciones pero no en otras. Las más comunes son las mutaciones
termosensibles en las que el producto del gen mutado funciona de modo normal a
una temperatura, la temperatura permisiva, pero no a otra, llamada restrictiva.
Enla mayoría de los casos, las mutaciones termosensibles son permisivas a
20-25°C y restrictivas a 35-37°C. De entre todos los mutantes termosensibles
obtenidos se seleccionaron aquellos que afectaban a un gen del control del
ciclo celular y que se diferenciaban del resto porque todas las células de una
población que portan esa mutación se paran en el mismo punto del ciclo celular cuando
se incuban a la temperatura restrictiva. A estas mutaciones, y por
extrapolación a los genes a los que afectan se les llama cdc -Ciclo de División
Celular-.
Estos
estudios han permitido identificar una familia de proteínas quinasas dependientes
de ciclina, -Cyclin Dependent Kinases, Cdk- que incluyen a Cdc28 de S.
cerevisiae, y a su homólogo en S. pombe cdc2, y que juegan un papel central en los
procesos claves del ciclo celular: la entrada en ciclo, la síntesis de ADN y la
regulación de la mitosis. Estas quinasas son las subunidades catalíticas de un complejo
que incluye tambiénuna subunidad reguladora, denominada ciclina, necesaria para
la función del complejo al determinar la localización o la especificidad de
sustrato de la quinasa. Esta asociación de subunidad catalítica y reguladora
define las funciones de la quinasa a lo largo del ciclo. En S. cerevisiae existen
al menos 9 ciclinas que se asocian a Cdc28. Por su estructura se dividen en dos
familias: ciclinas Cln –de Ciclina - que incluyen a Cln1, Cln2 y Cln3, que se necesitan
para el paso de la célula por el punto de Inicio presente en G1, y las ciclinas
Clb – de Ciclina tipo b - que incluyen a las seis restantes, Clb1-6, que son esenciales
para la regulación de la entrada en fase S y en Mitosis.
En concreto, cada
complejo Cdc28/Ciclina determina una función de la quinasa a lo largo del ciclo
(figura 5). Los complejos Cdc28/Ciclinas Cln activan en G1 el paso por Inicio,
la aparición de la yema y la degradación de Sic1, un inhibidor de los complejos
quinasa Cdc28/Clb [Nota 6], de modo que inactivan la capacidad de las células
haploides de pararse en G1 como respuesta a las feromonas sexuales, lo que proporciona
el carácter de irreversibilidad a la entrada en el ciclo celular. Los complejos
Cdc28/Ciclinas Clb activan el inicio de la replicación, regulan la formación de
los husos mitóticos y la división nuclear y el crecimiento isométrico de la
yema en G2 y M.
En los últimos años se han encontrado también homólogos a
estas quinasas en todos los eucariotas en los que se han buscado, y que
incluyen desde el gusano nematodo Caenorhabditis elegans, a la mosca Drosophila
melanogaster , a mamíferos como el ratón y el hombre y plantas como Arabidopsis
thaliana, indicando que el sistema de control del ciclo celular es general en
todos los eucariotas y validando a las levaduras como modelos de estudio.
El control de la fase
S y el inicio de la replicación
Dentro del sistema de control central que desencadena los
procesos esenciales del ciclo celular existen dispositivos bioquímicos que
actúan para poner en marcha un proceso que inicia la fase S.
¿Cómo se inicia?
Numerosos estudios acerca de los mecanismos de replicación del material
genético y de su regulación han ido conduciendo progresivamente a la descripción
de una serie de factores necesarios para su inicio y control. El Modelo Ciencia al Día © Febrero 2000, Vol. 3, No. 1.
ISSN 0717-3849 http://www.ciencia.cl/CienciaAlDia/volumen3/numero1/articulos/articulo3.html
8 del Replicón, inicialmente propuesto
para la bacteria Escherichia coli -E. coli - (Bell et al., 1993) predice la
existencia de secuencias de ADN desde donde se inicia la replicación.
Posteriormente estas secuencias fueron identificadas en S. cerevisiae y se
conocen como Secuencias de Replicación Autónoma (SRA) [Nota 7], las cuales
coinciden con los orígenes de replicación. Los cromosomas de eucariotas son
demasiado largos para ser replicados a partir de un solo punto como ocurre en procariotas,
por lo que contienen varios orígenes de replicación. Los trabajos de Bell y
Stillman de 1993 describieron un complejo de 6 proteínas que reconocen los orígenes
de replicación, denominado Complejo de Reconocimiento del Origen (CRO). Estas
proteínas son esenciales para la viabilidad celular y necesarias, pero no
suficientes, para iniciar la replicación del ADN. Otras proteínas iniciadoras
se han ido descubriendo paulatinamente, del tipo de Cdc6, las seis de la
familia Mcm, la quinasa Cdc7 - y su ciclina Dbf4 - y Cdc45.
Las proteínas CRO
permanecen unidas a los orígenes de replicación durante todo el ciclo celular,
pero el resto de las proteínas iniciadoras forman o no parte del complejo
dependiendo de la fase del ciclo, definiendo así un estado post-replicativo del
origen presente durante las fases S, G2 y M (sólo permanecen unidas a los orígenes
las proteínas CRO) y un estado pre-replicativo durante G1 (con el resto de las
proteínas iniciadoras formando parte del complejo). El inicio de la replicación
del material genético marca la transición entre el estado de complejo
pre-replicativoCoordinación entre el inicio de la fase S y el ciclo celular .
Las evidencias que se
tenían acerca de la necesidad de actividad Cdc28/Clb para que ocurriese la
síntesis del material genético (Schwob & Nasmyth, 1993), contrastaban con
el hecho, posteriormente descubierto (Dahmann et al., 1995), de que la
formación de los complejos pre-RC, absolutamente necesarios para que dicho
proceso se inicie, se inhibía en presencia de actividad quinasa. Esta aparente paradoja
no es más que el reflejo del exquisito control que, sobre los diferentes sucesos
del ciclo celular, ejercen las oscilaciones en actividad quinasa a lo largo del
mismo.
De una parte, la
fosforilación dependiente de Cdc28/Cln de factores de transcripción específicos
de genes de fase S controla la transcripción [Nota 8] de genes necesarios para
la replicación, pero son las ciclinas tipo B, Clb5 y Clb6, las que activan
directamente la replicación del ADN. Por otro lado, la conversión de complejos
post-RC a pre-RC en los orígenes ocurre en la transición M/G1 cuando la
actividad quinasa Cdc28/Clb es baja y la conversión recíproca, en la transición
G1/S coincidiendo con la elevación de la actividad quinasa debida a los
complejos Cdc28/Clb (figura 7).
La unión y la
separación de todos los componentes que constituyen los pre-RC se verían
reguladas por procesos de desfosforilación-fosforilación y/o localización subcelular
mediados por los complejos Cdc28/Clb.
La actividad quinasa asociada a complejos de Cdc28/Clb
aparece al comienzo de la fase S y aunq pueden detectarse complejos Cdc28/Clb5
y Cd28/clb6 ya en fase G1 , estos son inicialmente inactivos debido a la
presencia de sicl , quien regula la entrada de la fase S regulando la actividad
quinasa de los complejos Cdc28/Clb5 y
Cdc28/clb6. La función principal de los complejos Cdc28/cln1 yCdc28/cln2
en promover fase S parece ser la degradación de sic1 poco antes del comienzo de
fase S
Se ha propuesto un modelo de inicio de la replicación en dos
fases sucesivas e interdependientes :primero tendría lugar el estableciminento
de los complejos preRC en presencia de Cdc6 y con el reclutamiento de la
proteínas Mcm y Cdc45 , y después se iniciaría la replicación del ADN promovido
por la actividad quinasa debida tanto a Cdc28/Clb como a Cdc7/Dbf4 , con el
siguiente desmontaje de los pre-RC (figura 8 )
Mantenimiento de la
ploidía [Nota 9]
Si una célula inicia la replicación de su material genético
más de una vez antes de dividirse, se encuentra con serios problemas a la hora
de repartirlo durante la mitosis, de modo que las dos células resultantes
tienen cantidades de material hereditario distintas entre sí y desiguales a la
célula que inició ese ciclo de división celular. Semejantes defectos serían
arrastrados en divisiones sucesivas haciendo inviables a las células
resultantes Parece vital, por tanto, llevar a cabo una replicación fidedigna y
única por ciclo para que se mantenga la ploidía durante las sucesivas
divisiones proliferativas de una célula
El modelo de regulación del inicio de la replicación en dos
pasos garantiza a la célula el mantenimiento de su ploidía. Si la condición
necesaria para que los componentes requeridos para la formación de los
complejos pre-RC se ensamblen es la ausencia de actividad quinasa su presencia
se requiere para la activación de dichos complejos y el inicio de la
replicación. La subida de actividad quinasa en la transición G1/S (favorecida
por la destrucción de proteínas inhibidoras de
Cdc28/Clb) a la vez dispara el inicio de la replicación en
los orígenes con la consecuencia del
sesensamblaje de los propios compleos pre-RC cuyo reensamblaje queda
inhibido hasta una nueva fase G1 después de la bajada de actividad quinasa que
marca la salida de la mitosis y por tanto después de que el material genético
previamente duplicado haya sido adecuadamente repartido. El mismo tipo de
molécula (Cdc28/Clb) tiene un papel dual: una función positiva para permitir
una función legítima (activación de los orígenes e inicio de la replicación del
ADN en fase S) y una negativa, para impedir una función ilegítima (bloqueo del ensamblaje
de nuevos complejos pre-RC durante las fases S, G2 y M). Cuando la actividad
legítima ha sido ejecutada, se activa la ilegítima
Lo que queda por
saber
Afortunadamente para los estudiosos de esta materia, el
asunto no está· zanjado. Aunque la actividad Cdc28 es decisiva en la regulación
de la formación de los complejos pre-RC,
sus substratos y el efecto de las fosforilaciones sobre ellos para dirigir
su actividad, destrucción o localización subcelular, son poco conocidos. Los diferentes
componentes de los complejos del origen son los candidatos de primera línea
para establecer el nexo (Cdc6, Mcms, CRO). De hecho, ya se ha descrito la fosforilación
por Cdc28 de varias de estas moléculas (Dutta & Bell, 1997) y existen evidencias
recientes de la existencia de procesos de re-localización subcelular de moléculas
claves para el control del ciclo celular. Esta última estrategia permite sacar
del escenario al actor una vez representado su papel, con el fin de que su presencia
no impida ni pasar al acto siguiente ni ejercer su papel a otro protagonista.
Así, además de los mecanismos ampliamente descritos de destrucción proteolítica
[Nota 10] o inhibición directa por otra proteína, ciertos actores pueden ser
eliminados de escena mediante su secuestro en un escenario celular distinto al de
su lugar de actuación (citoplasma, nucleolo). Este atractivo mecanismo ha sido descrito
para las proteínas Mcm (Dutta & Bell, 1997).
El descubrimiento de
los cambios que se producen en la cromatina para hacer posible el acceso de la
maquinaria de replicación al ADN, así como todos los factores responsables de
la elongación de las hebras de nueva síntesis, una vez iniciada ésta, vendrían
a completar el escenario de uno de los procesos fundamentales en la vida
celular: la replicación del ADN.
Notas
1. La información genética de un organismo se encuentra
contenida en unas estructuras denominadas cromosomas, compuestos por una
molécula de ADN y por proteínas asociadas, especialmente evidentes en células
en mitosis o meiosis.
2. La molécula portadora de la información genética es el
ácido desoxirribonucleico (ADN) que es un polinucleótido formado por la unión
covalente de desoxirribonucleótidos en formando una estructura bicatenaria en
hélice.
3. Los organismos
eucariotas incluyen tanto seres unicelulares como pluricelulares que se
caracterizan por presentar en el citoplasma un núcleo diferenciado que alberga
el material genético. Por el contrario, los organismos procariotas están constituidos
por células sencillas que carecen de núcleo verdadero (bacterias y cianobacterias).
4. Cualquier
eliminación de una porción de secuencia de una molécula de ADN, se conoce con
el término de deleción. En este contexto se refiere a la eliminación completa
de un gen, lo que constituye una herramienta muy útil en el análisis de las funciones
de la proteína para la que codifica ese gen. Aquellas funciones ausentes en la
célula delecionada para un gen, permiten deducir qué función tiene su producto
génico.
5. Los organismos eucariotas pueden tener un juego completo
de cromosomas (organismos haploides), dos juegos (organismos diploides como es
el caso humano que cuenta con 23 parejas de cromosomas), cuatro (organismos tetraploides)
e incluso más.
6. La regulación de la actividad quinasa de los complejos
Cdc28/ciclina en la célula es muy precisa, dada su importancia. Esta regulación
tan fina se logra modulando la actividad quinasa a varios niveles: se puede
regular la subunidad catalítica Cdc28 (mediante
fosforilaciones-desfosforilaciones activadoras o inhibidoras), la presencia de
las ciclinas (controlando su transcripción y su degradación específica), y el
complejo (mediante proteínas que inhiben su función, como Sic1 en S.
cerevisiae).
7. S. cerevisiae es el único eucariota en el que se conocen
en detalle las SRA. Una típica, consta de varios elementos. El primero,
conocido como elemento A, está formado por una secuencia de 11 pares de bases,
en su mayoría de Adeninas y Timinas. Adicionalmente aparecen hasta 3 elementos
más, B1, B2 y B3, adyacentes al A y que se extienden a lo largo de 150
nucleótidos. Esta composición de elementos varía de un SRA a otro, pudiendo
haber elementos repetidos e incluso carecer de uno o varios de ellos.
8. El ADN alberga la información genética, necesaria para
que la célula sepa como
realizar cada proceso celular. Sin embargo carece de
actividad catalítica. Para que esos procesos tengan lugar esa información debe
“traducirse” a moléculas que sí tengan actividad catalítica, las proteínas.
Este proceso de transmisión de información consta de un paso intermedio, la
transcripción: el ADN es copiado a ARN mensajero, que transporta esa
información al citoplasma, que es donde radica en la célula la maquinaria de
traducción.
9. El término ploidía
hace referencia al número de juegos cromosómicos de las células de un
organismo.
10. Una de las formas
de asegurar la irreversibilidad de un proceso bioquímico es la destrucción de
un participante necesario para que ese proceso tenga lugar. En S. cerevisiae
existe un complejo de proteínas codificadas por los genes CDC4, CDC34 y CDC53
que controlan la transición G1/S mediante la degradación de proteínas marcadas
con ubiquitina (King et al., 1996). Es el caso de la degradación de Cdc6 (Sánchez
et al., 1999) que asegura que una vez iniciada la síntesis de ADN, el proceso
no vuelva a ocurrir ya que Cdc6 es necesaria para formar nuevos pre-RC.
Bibliografía
Hartwell, L. H., Culotti, J. & Reid, B.
(1970). Genetic control of cell division in yeast, I. Detection of mutants.
Proc. Natl. Acad. Sci. USA 66:352-359. Nurse, P., Thuriaux, P. & Nasmyth,
K. (1976). Genetic control of the cell division cycle in the fission yeast
Schizosaccharomyces pombe. Mol. Gen. Genet. Jacob, F., Brenner, S. & Cuzin,
F. (1963). On the regulation of DNA replication in bacteria. Cold Spring Harbor
Symp. Quant. Biol. 28, 383-387. Bell, S. P., Kobayashi, R. & Stillman, B.
(1993). Yeast origin recognition complex functions in transcription silencing
and DNA replication. Science 262, 1844-1849.Schwob, E. & Nasmyth, K.
(1993). CLB5 and CLB6, a new pair of B cyclins involved in DNA replication in Saccharomyces cerevisiae.
Genes Dev. 7, 1160-1175.
Dahmann C.,
Diffley, J. F. X. & Nasmyth K.A. (1995). S-phase-promoting cyclin dependent
kinases prevent re-replication by inhibiting the transition of origins to a
pre-replicative state. Curr. Biol. 5,
1257-1269.Dutta, A. & Bell, S. P. (1997). Initiation of DNA replication in
eukaryotic cells. Annu. Rev. Cell Dev. Biol. 13, 293-332. King, R. W.,
Deshaies, R. J., Peters, J. M. & Kirschner, M. W. (1996). How proteolysis
drives the cell cycle. Science 274,
1652-1659. Sánchez, M., Calzada, A. & Bueno, A. (1999). The Cdc6 protein is
ubiquitinated in vivo for proteolysis in Saccharomyces cerevisiae. The Journal
of Biological Chemistry 274: 9092-
No hay comentarios:
Publicar un comentario